Un regalo di Natale per L.U.C.A.
Caro L.U.C.A.,
Natale é già passato da quasi due settimane, ma trovo solo ora il tempo di mandarti una foto del regalo che ho deciso di farti per Natale! Come ben sai, tu sei l’ultimo antenato comune tra tutte le specie viventi, il nostro Last Universal Common Ancestor, e per questo ho deciso di regalarti un pesce! Mi prenderò cura di lui come se fosse un mio parente (anche se effettivamente lo é, perché risalendo i nostri alberi genealogici fino a 3,6/4 miliardi di anni fa dovremmo trovare entrambi te) e posterò regolarmente delle foto e dei dati sulla sua crescita.
Facendo delle ricerche sul tuo conto ho scoperto che in fondo, ma molto in fondo, ho dei legami, per quanto lontani, con tutti gli esseri viventi. Mi prenderò quindi cura non solo del pesce, a cui ho dato il nome Sushi, ma anche dell’ambiente in cui lui vivrà (sempre che vivrà, visto che, come puoi vedere dalla foto, il mio gatto lo sta già puntando) e mi preoccuperò di adattare l’acquario alle sue esigenze.
Ho infatti in programma di portare presto degli amici pesci a far compagnia a Sushi, magari prendendogli anche una nuova casa.
Nel frattempo terrò a bada il nemico!
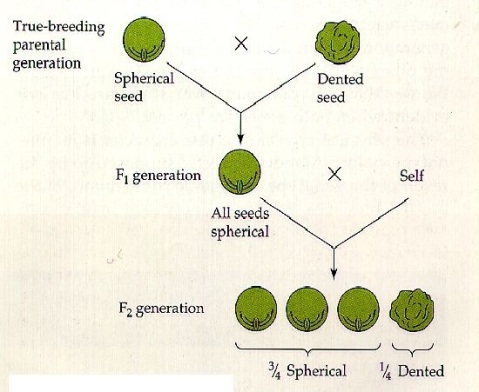
Quell’imbroglione di Mendel
Non tutti sanno che il geniale frate Gregor Mendel, per divenire quello che noi ora riteniamo il padre della genetica, imbrogliò. Viste le conoscenze e gli strumenti a disposizione del naturalista in quella lontana epoca storica, si ritiene sia impossibile, o comunque alquanto improbabile, che con così pochi dati da prendere in considerazione, i risultati rispecchiassero alla perfezione la sua teoria.
Ma non concentriamoci su come arrivò alle sue leggi, bensì su come quest’ultime sono e che cosa comportano.
Ecco una dettagliata spiegazione di come si svolse il lavoro del frate agostiniano.
Le mutazioni geniche
Per mutazione genica si intende quella variazione, anche minima, che avviene nella struttura molecolare di un gene e che può riguardare un solo nucleotide, come anche più nucleotidi, della catena di DNA, come ad esempio la sostituzione di una purina o di una pirimidina con un’altra base azotata (per es.: come avviene nell’anemia falciforme si ha la sostituzione della timina con l’adenina). Una volta avvenuta questa mutazione nella struttura molecolare del materiale genico, essa si riproduce a ogni replicazione del DNA e cioè diventa ereditaria; soltanto un altro cambiamento potrà produrre una nuova mutazione che potrebbe anche consistere in un ritorno alla struttura originale.
Sono, quindi, le mutazioni che alterano un singolo gene e dunque le più “piccole” che si possono avere. In quanto tali non sono visibili attraverso analisi al microscopio (tranne alcuni casi estremi), ma possono essere riscontrate solo tramite analisi genetiche. Le mutazioni geniche portano alla formazione di nuove forme geniche, ovvero di nuovi alleli, detti appunto alleli mutanti. In quanto tali questi sono rari nella popolazione e si differenziano dagli alleli più diffusi detti invece tipi selvatici. Bisogna però far distinzione anche tra alleli mutanti e morfi. I morfi sono infatti due o più alleli di uno stesso gene con frequenza superiore all’1% (polimorfismo). Alla luce di questo ne deriva che il concetto di mutazione non è assoluto: un gene potrà subire una mutazione; se l’allele mutante però troverà le condizioni per diffondersi nella popolazione e superare la frequenza dell’1% non si parlerà più di mutazione ma di morfo.
Possono essere distinte in due categorie: mutazioni puntiformi e mutazioni per sequenze ripetute. Le prime sono causate da sostituzioni di basi o da inserzioni o delezioni di coppie di basi (mutazioni indel). La seconda categoria comprende le mutazioni causate sempre da inserzioni o delezioni ma di sequenze di basi ripetute.
Mutazioni puntiformi
Sostituzioni di basi: Determinano uno scambio di un nucleotide con un altro. Sono definite transizioni qualora vi è un scambio di una purina con altra purina (A ↔ G) o di una pirimidina con un’altra pirimidina (C ↔ T); oppure transversioni quando lo scambio è di una purina con un a pirimidina o viceversa (C/T ↔ A/G). In genere le transizioni sono più frequenti delle transversioni. Quando ci si riferisce a mutazioni di una sequenza che codifica per un determinato prodotto genico le sostituzioni potranno essere:
Mutazioni per sequenze ripetute
Analoghe alle mutazioni indel, interessano però più di un nucleotide adiacente; in particolare interessano gruppi nucleotidici che formano una sequenza la quale si ripete più volte di seguito. La mutazione, che si origina nel corso della replicazione del DNA, provoca una variazione nel numero di queste sequenze ripetute; il nuovo filamento di DNA potrà presentarne in eccesso o in difetto. Il fenomeno che causa la mutazione è detto slittamento della replicazione (replication slippage). Malattie genetiche associate a questo tipo di mutazione sono la Corea di Huntington e la sindrome dell’X fragile.
Effetti delle mutazioni geniche
Gli effetti possono essere notevolmente diversi a seconda del tipo di mutazione e della posizione in cui questa si verifica. Una mutazione può non portare a nessuna conseguenza e questo quando interessa DNA che non codifica (o meglio sembra non codificare) per nessun prodotto genico (il cosiddetto junk DNA o DNA spazzatura). Se la mutazione va invece ad alterare le sequenze codificanti, ovvero i geni, si ha una variazione nel tipo o nella quantità del corrispettivo prodotto genico, che può essere una proteina o RNA funzionale (rRNA, tRNA, snRNA ecc.). Parliamo in questo caso di mutazione biochimica; se la mutazione biochimica porta a una variazione visibile del fenotipo si parla di mutazione morfologica.
Inoltre distinguiamo, sempre in relazione agli effetti, in:
mutazione positiva: quella che porta un vantaggio evolutivo;
mutazione neutra: quella che non risulta in un depotenziamento della capacità riproduttiva dell’individuo;
mutazione subletale: quella che rende più difficoltosa la perpetuazione riproduttiva dell’individuo (il tipico esempio sono le malattie genetiche che debilitano in qualche modo l’individuo, rendendolo meno capace di riprodursi, senza però impedirglielo totalmente);
mutazione letale: quella che non permette all’individuo di raggiungere l’età riproduttiva o non gli permette di riprodursi.
L’efficacia della mutazione, sia positiva che negativa, dipende poi dal tipo di allele mutato così creato; questo potrà essere infatti dominante o recessivo. Nei diploidi (2n cromosomi) se è dominante avrà sempre effetto (sia in un eterozigote che in un omozigote dominante); se è recessivo, essendo aploinsufficiente, per avere effetto ha bisogno che anche l’altro elemento della coppia genica sia mutato (individuo omozigote recessivo). Negli aploidi , che sono emizigoti (n cromosomi), la mutazione avrà invece sempre effetto. (Fonte adattata e rielaborata: ftp://89-97-218-226.ip19.fastwebnet.it/web1/DNA/dna.htm)
Dal minuto 4.47 del video, un esempio di quotidiana mutazione genica:
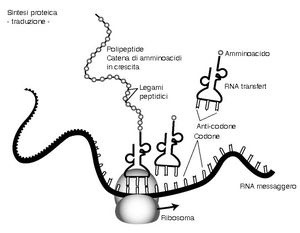
La sintesi proteica
La traduzione è lo stadio della sintesi proteica in cui le istruzioni portate dall’m-RNA vengono tradotte nella sequenza corretta di amminoacidi per formare una proteina.
La traduzione ha luogo nel ribosoma (formato da r-RNA e proteine), composto da due subunità: quella piccola contiene un sito di legame per l’m-RNA; quella grande ha due siti di legame per due molecole di t-RNA e un sito che catalizza la formazione del legame peptidico tra due amminoacidi adiacenti.
Ogni molecola di t-RNA è specifica per un unico amminoacido ed è in grado di riconoscere sia l’amminoacido che deve trasportare, sia il codone complementare di m-RNA associato al ribosoma.
La traduzione ha inizio quando due codoni del filamento di m-RNA si legano alla subunità piccola di un ribosoma. Il primo codone è la tripletta di “inizio lettura” AUG, alla quale corrisponde l’amminoacido metionina; il secondo codifica il primo vero amminoacido della proteina. I due t-RNA, che hanno rispettivamente l’anticodone di inizio e l’anticodone complementare al secondo codone, si legano alla subunità grande e si forma un legame peptidico (cioè il legame tra amminoacidi che forma le proteine) tra i due amminoacidi trasportati.
Il t-RNA di inizio si stacca dal ribosoma mentre il dipeptide (i due amminoacidi uniti dal legame peptidico) rimane legato al secondo t-RNA. Il ribosoma si sposta sopra un altro codone dell’m-RNA e una nuova molecola di t-RNA con il proprio amminoacido si dispone nel sito di legame vuoto del ribosoma. Si crea un nuovo legame peptidico e il tripeptide si salda all’ultimo t-RNA. Il processo di allungamento della catena polipeptidica prosegue in questo modo finché tutte le triplette sono state tradotte e viene raggiunto il codone di “fine lettura”. La proteina completa si stacca dal ribosoma e specifici enzimi scindono il legame con la metionina.
Ecco inoltre un video esemplificativo dell’argomento trattato:
Il codice genetico
Come ben sappiamo ogni cromosoma é formato sia da proteine che da DNA. Ogni proteina contiene 20 amminoacidi differenti, ma il DNA e l’RNA, fonti del codice genetico delle proteine, contengono solo quattro basi azotate. Come é possibile?
Se ciascun nucleotide codificasse per un amminoacido, alle quattro basi corrisponderebbero solo quattro amminoacidi. Analogamente, se un amminoacido fosse codificato da due nucleotidi ci sarebbero al massimo 16 amminoacidi, comunque non sufficienti. Per ottenere un numero più che sufficiente di combinazioni, ogni amminoacido deve essere quindi codificato da tre nucleotidi, per un totale di 64 amminoacidi. Ogni combinazione é quindi costituita da una sequenza di tre nucleotidi chiamata codone.
Di queste 64 combinazioni, 3 rappresentano segnali di arresto (UUA, UAG e UGA) e le restanti 61 dei veri e propri amminoacidi, corrispondenti ai soli 20 presenti nella proteina. Ciò é possibile poiché i codoni ai quali corrisponde uno stesso amminoacido differiscono spesso solo per il terzo nucleotide ed é per questo che il codice viene definito degenerato.
Un simpatico processo chiamato Splicing
Molti geni negli eucarioti superiori codificano per RNA che possono essere tagliati in modi diversi (persino migliaia come in Drosophila) per generare due o più RNA differenti e, di conseguenza, diversi prodotti proteici in un processo detto appunto splicing alternativo. Difatti, oltre alla scelta di esoni alternativi, alcuni esoni possono essere estesi ed altri addirittura saltati (di proposito). Inoltre, alcuni introni possono essere mantenuti, anziché eliminati, e dar luogo perciò a prodotti proteici ancora differenti. Di conseguenza, si può dire che lo splicing alternativo può essere sia costitutivo, sia regolato. Nel primo caso, più di un prodotto viene sempre generato dal gene trascritto. Nel caso dello splicing regolato, forme diverse vengono generate in momenti diversi, in diverse condizioni o in diversi tipi cellulari o tessuti. Comunque, le proteine che regolano lo splicing si legano a dei siti specifici chiamati enhancer o silenziatori di splicing esonici o intronici (exonic – intronic – splicing enhancers o silencers, ESE o ISE – ESS o ISS). I primi incrementano mentre i secondi reprimono lo splicing sui siti di splicing vicini. Abbiamo già incontrato gli enhancer e le proteine SR che si legano ad essi, portando il macchinario di splicing su diversi siti. Perciò, la presenza o l’attività di una data proteina SR può determinare quale sito particolare di splicing venga utilizzato in un determinato tipo cellulare o in un particolare momento dello sviluppo. La maggior parte dei silenziatori, invece, vengono riconosciuti dai membri della famiglia delle ribonucleoproteine nucleari eterogenee (hnRNP, heterogeneous nuclear ribonucleoprotein). Quest’ultime legano l’RNA ma non hanno nessun dominio che riesca a far reclutare il macchinario di splicing e quindi impediscono l’utilizzo di siti specifici di splicing. Un altro repressore dell’ splicing di mammifero è la proteina hnRNP, che blocca il legame del macchinario di splicing attraverso il suo legame con la regione polipirimidinica. In generale, si considera lo splicing alternativo come un modo per produrre proteine diverse da un singolo gene. Queste proteine diverse sono chiamate isoforme. Possono avere funzioni simili, funzioni distinte e persino funzioni antagoniste. Ma anche alcuni geni che codificano per una sola proteina funzionante subiscono uno splicing alternativo. In questi casi, questo meccanismo viene usato semplicemente per spegnere o accendere l’espressione del gene. Infatti, lo splicing alternativo, in quest’ultimo caso determina se l’esone con il codone di stop venga incorporato nell’mRNA e, di conseguenza, se il gene venga espresso o no. La seconda maniera in cui lo splicing alternativo viene usato per l’accensione o lo spegnimento di un gene è attraverso la regolazione dell’utilizzo di un introne, il quale, se mantenuto nell’mRNA, impedisce all’mRNA stesso di essere esportato dal nucleo e perciò tradotto. (Tratto da tesionline.it)
RNA: il processo di trascrizione
Le molecole di mRNA sono assemblate partendo da un filamento di DNA, in base allo stesso principio di appaiamento delle basi azotate che regola la duplicazione del DNA. Questo processo di sintesi dell’RNA è definito “trascrizione” e ha la funzione di trascrivere il messaggio contenuto di un frammento di DNA in un trascritto di RNA.
I siti di legame per la RNA polimerasi sono detti promotori e si trovano sul filamento di DNA da trascrivere, come anche le sequenze d’arresto, sequenze nucleotidiche che bloccano la sintesi dell’RNA.
L’mRNA è una molecola che ha il compito di trasportare le informazioni del DNA all’interno del citoplasma.
Il Processo di trascrizione è composto da tre fasi:
-inizio: l’RNA polimerasi riconosce il promotore e si attacca ad esso;
-allungamento: l’RNA polimerasi sintetizza il trascritto di mRNA, partendo da un filamento di DNA detto filamento stampo;
-terminazione. l’RNA polimerasi si incontra con la sequenza di arresto e il processo di trascrizione termina.
DNA, Hot Pockets, & The Longest Word Ever
Sempre dal canale youtube crashcourse, ecco un fantastico video (in inglese e con i sottotitoli in inglese) che riassume in 18 minuti tutto quello che abbiamo fatto in biologia nel corso delle ultime settimane!
Dalla trascrizione del DNA alla struttura delle proteine, passando per la parola più lunga del mondo: il nome di una proteina!
Per comodità sono qui sotto elencati gli argomenti trattati nel video:
1) Transcription 2:12
A) Transcription Unit 3:00
B) Promoter 3:10
C) TATA Box 3:32
D) RNA Polymerase 4:12
E) mRNA 4:15
F) Termination signal 5:21
G) 5′ Cap & Poly-A Tail 5:34
2) RNA Splicing 6:08
A) SNuRPs & Spliceosome 6:26
B) Exons & Introns 6:56
3) Translation 7:28
A) mRNA & tRNA 8:01
B) Triplet Codons & Anticodons 8:39
4) Folding & Protein Structure 10:51
A) Primary Structure 11:11
B) Secondary Structure 11:23
C) Tertiary Structure 11:58
D) Quaternary Structure 12:44
PCR- Reazione a catena della polimerasi
La reazione a catena della polimerasi è una tecnica che consente la moltiplicazione di frammenti di DNA dei quali si conoscano le sequenze nucleotidiche iniziali e terminali. L’amplificazione mediante PCR consente di ottenere molto rapidamente la quantità di materiale genetico necessaria per le successive applicazioni. La PCR viene utilizzata in tutte quelle situazioni in cui bisogna amplificare un quantitativo di DNA fino a livelli utili per analisi successive. I campi di applicazione sono enormi. La tecnica viene sfruttata, per esempio, in medicina per la diagnostica microbiologica o per l’evidenziazione di cellule tumorali, in tumori liquidi, quando esse sono troppo poche per essere evidenziate da altre metodiche (malattia minima residua). Estremamente utile è l’uso della PCR in medicina legale. In biologia la PCR viene usata per le analisi di paleontologia e di antropologia molecolare ed in numerosi campi dell’ingegneria genetica. (Definizione adattata da Wikipedia)
Ecco come funziona la PCR, con una breve introduzione sulla struttura del DNA che termina al minuto 2.34:
Il Proofreading
Nel processo di duplicazione del DNA non sono assenti errori, che, se non corretti, potrebbero portare a gravi conseguenze. Per nostra fortuna esiste il proofreading, quel processo che consente il riconoscimento e la correzione di errori nel DNA.
Durante la replicazione, un gruppo di enzimi tiene costantemente sotto controllo la doppia elica del DNA e quando incontra un errore (errato accoppiamento di nucleotidi o basi azotate) può operare in diversi modi:
-RIPARAZIONE DIRETTA: l’enzima riconosce l’errore nel DNA e lo corregge direttamente;
-RIPARAZIONE PER TAGLIO DI BASI/NUCLEOTIDI: la porzione di filamento con una base o un nucleotide sbagliato viene rimossa, il filamento di DNA complementare viene usato come stampo per sintetizzare la nuova porzione corretta;
RIPARAZIONE DI UN APPAIAMENTO ERRATO: questo sistema è simile al precedente, ma l’errore è nell’appaiamento delle basi; quando l’errore viene riconosciuto, la porzione è rimossa e il filamento di DNA complementare viene usato come stampo per sintetizzare la nuova porzione corretta.
Definizione dei metodi di riparazione liberamente tratta dal libro di testo “Invito alla biologia.blu, Zanichelli”
La duplicazione del DNA
Una delle caratteristiche fondamentali del materiale genetico é quella di produrre copie identiche di se stesso. Ciò è possibile grazie alla struttura a doppia elica del DNA, ma come funziona questa autoriproduzione?
Il sito liceogalileogalilei.it ci fornisce una semplice presentazione per incominciare a capire come funziona questo complicato processo.
Per chi ha maggiore sete di sapere, ecco altre utili fonti per lo studio:
Quest’altro video é invece molto chiaro e ben fatto, se non fosse per la voce robotica che spiega il processo:
Questo filmato, della durata di quasi mezz’ora, é più completo e complicato, per chi vuole approfondire ancora di più sull’argomento:
La struttura del DNA e del RNA
Dal sito della DeAGOSTINI sapere.it, ecco un utile riassunto delle nozioni riguardante la struttura del DNA e del RNA.
I Nucleotidi e le Basi Azotate
Già all’inizio del ‘900 era noto che il DNA é costituito da nucleotidi. Ogni nucleotide é composto da una base azotata, da uno zucchero a 5 atomi di carbonio (deossiribosio) e da un gruppo fosfato.
Le basi azotate sono quattro e si suddividono in due gruppi:
-purine (adenina e guanina), che presentano una struttura a due anelli;
-pirimidine (citosina e timina), che presentano una struttura ad un solo anello.
La doppia elica del DNA, che per comodità può essere immaginata come una scala a chiocciola, é costituita da dei “pioli” che possono essere formati solo dalle coppie adenina-timina e guanina-citosina.
Gli enzimi
Gli enzimi sono i catalizzatori dei sistemi biologici. […] La stragrande maggioranza degli enzimi sono proteine (proteine enzimatiche). Una piccola minoranza di enzimi sono molecole di RNA. Le molecole di RNA dotate di potere catalitico costituiscono una sottocategoria peculiare degli enzimi chiamata ribozimi (o enzimi a RNA). Il processo di catalisi indotto da un enzima (come da un qualsiasi altro catalizzatore) consiste in un’accelerazione della velocità della reazione e quindi in un più rapido raggiungimento dello stato di equilibrio termodinamico. Un enzima accelera unicamente le velocità delle reazioni chimiche, diretta e inversa (dal composto A al composto B e viceversa), senza intervenire sui processi che ne regolano la spontaneità.
Il suo ruolo consiste nel facilitare le reazioni attraverso l’interazione tra il substrato (la molecola o le molecole che partecipano alla reazione) e il proprio sito attivo (la parte di enzima in cui avvengono le reazioni), formando un complesso. Avvenuta la reazione, il prodotto viene allontanato dall’enzima, che rimane disponibile per iniziarne una nuova. L’enzima infatti non viene consumato durante la reazione.
Definizione liberamente tratta da Wikipedia
Ma come funzionano gli enzimi?
Ecco un video in inglese che lo spiega:
ANIMAZIONI: La storia del DNA, da Friedrich Miescher fino a Watson e Crick
Da quanto Friedrich Miescher, giocando con del pus, scoprì il DNA, da lui chiamato nucleina, iniziò una serie di esperimenti e ricerche che portò, nel 1953, alla scoperta della forma a doppia elica. Nel mezzo diverse geniali intuizioni portarono a scoprire che le informazioni genetiche non erano portate dalle proteine, ma proprio dal DNA.
Dal sito Dna From The Beginning, alcune animazioni su queste interessanti scoperte:
DNA: What is it? How does it work?
Navigando nel web ho scoperto un utilissimo e molto ben curato canale video di youtube, ovviamente in inglese.
Proprio qui ho trovato un interessante video spiegazione su cos’è il DNA e come funziona, con anche i sottotitoli in inglese per chi, come me, non è un abile ascoltatore:
Il DNA
Il DNA, o acido desossiribonucleico, è cio che contiene le informazioni genetiche dell’organismo.
Nel corso dell’ultimo secolo sono avvenute innumerevoli scoperte in merito a ciò e in occasione del cinquantenario della sua scoperta, avvenuto nel 2003, il Cold Spring Harbor Laboratory ha creato un sito contenente diverse informazioni accessibili a tutti sul DNA, poi tradotto in italiano dall’Università di Padova.
Proprio nel sito in italiano è presente un’utile timeline sulle più importanti scoperte sul DNA effettuate in questi 50 anni.